УДК 577.21:575.22:581.6 doi: 10.21498/25181017.19.4.2023.291223
Н. Е. Волкова1,2*, Г. І. Сліщук1,2, О. О. Захарова1,
Т. Ю. Марченко2, В. І. Січкар2, Р. А. Вожегова2
1ТОВ «КОТЕКНА УКРАЇНА ЛІМІТЕД», вул. Люстдорфська дорога, 140А, м. Одеса, 65114, Україна,
*email: natalia.volkova@cotecna.com
2Інститут кліматично орієнтованого сільського господарства НААН, вул. Маяцька дорога, 24, смт Хлібодарське, Одеська обл., 67667, Україна
Мета. Визначити поліморфізм мікросателітних локусів QTLhotspotрегіону групи зчеплення 4, пов’язаного з толерантністю до посухи, у сортів нуту звичайного української селекції. Методи. Екстрагування та очищення ДНК з проростків ЦТАБметодом; полімеразна ланцюгова реакція; горизонтальний гельелектрофорез; визначення розмірів продуктів ампліфікації за допомогою програми «Image J». Результати. Встановлено алельні комбінації мікросателітних локусів ICCM0249, NCPGR127, TAA170, NCPGR21, TA130 і STMS11 QTLhotspotрегіону групи зчеплення 4 геному нуту. За локусами STMS11, NCPGR127 і NCPGR21, які виявилися неполіморфними в межах аналізованої вибірки сортів, детектовано по одному алелю; за ICCM0249 і TAA170 – по два; за TAA130 – три алелі, що вказує на їхню поліморфність. Висновки. Серед семи сортів нуту української селекції три мікросателітні локуси є неполіморфними, а саме: STMS11, NCPGR127 і NCPGR21. Ще три – поліморфними з двома алелями для локусів ICCM0249 і TAA170 і трьома для TAA130. За результатами аналізу сортів встановлено п’ять типів комбінацій алелів мікросателітних локусів ICCM0249, NCPGR127, TAA170, NCPGR21, TA130 і STMS11. У сорту ‘Пам’ять’ ідентифіковано унікальний для досліджуваної вибірки алель розміром 185 п. н.
Ключові слова: нут; толерантність до посухи; поліморфізм; мікросателітний локус; ПЛР.
Nataliia Volkova
https://orcid.org/0000000293334872
Heorhii Slishchuk
https://orcid.org/0000000342458557
Olga Zakharova
https://orcid.org/0000000318289044
Tetiana Marchenko
https://orcid.org/0000000169943443
Vyacheslav Sichkar
https://orcid.org/0000000305815068
Raisa Vozhehova
https://orcid.org/0000000238955633
Вступ
Підвищення толерантності основних зернових і бобових культур до дефіциту вологи для отримання стабільно високих врожаїв в умовах змін клімату є актуальним завданням селекції. Одна з її найефективніших стратегій – генно-інженерні технології, успішним прикладом яких є створення стійких проти посухи генномодифікованих подій кукурудзи, пшениці, цукрової тростини та сої [1–4]. Втім все ще насторожене ставлення до генетично модифікованих рослин, а також потреба генно-інженерних технологій в обладнанні та фахівцях високого рівня зумовлюють необхідність використання альтернативних методів маркерної селекції.
Нут (Cicer arietinum L.) – бобова культура, яку вирощують на всіх континентах. Він є цінним джерелом протеїнів, вітамінів і необхідних мінералів для харчування людини. Також його важлива функція полягає в підвищенні родючості ґрунту завдяки симбіотичній фіксації атмосферного азоту. Цю культуру вважають перспективною для України через високу ліквідність насіння на ринку [5].
Важливим етапом розроблення покращених, толерантних до майбутніх змін клімату сортів нуту є ідентифікація генів-кандидатів і діагностичних маркерів, пов’язаних з ключовими ознаками адаптації до посухи. Внаслідок процесів доместикації нут має вузьку генетичну базу, а тому знижується ефективність сучасних стратегій підвищення врожайності [6].
Здійснено секвенування геномів нуту типів «desi» лінії ‘ICC4958’ [7, 8] і «kabuli» лінії ‘CDC Frontier’ [9, 10]. Розмір одного геному – 738 Mb. Анотовано 28269 генів, розташованих у восьми гомологічних парах хромосом (2n = 16) [11]. Для кращого розуміння генетики нуту щодо стійкості проти посухи проведено широкомасштабні дослідження локусів кількісних ознак (англ. Quantitative Trait Locus, QTL) за використання різних типів молекулярних маркерів [12–16].
Зокрема, в групі зчеплення 4 геному нуту (CaLG04) ідентифіковано геномну область QTL-hotspot-регіон, що містить локуси для кількох ознак, пов’язаних із толерантністю до посухи, та відповідає за 58,2% фенотипових варіацій. Внаслідок інтрогресії QTL-hotspot-регіону в елітні сорти нуту індійської селекції з використанням маркер-опосередкованого беккросингу (англ. Marker-assisted backcrossing, MABC) врожайність насіння інтрогресованих ліній в умовах посухи збільшено на 16%, порівнюючи з рекурентними батьківськими генотипами, та покращено такі властивості коренів, як загальна довжина, щільність довжини, площа поверхні та об’єм. Також у цьому QTL-hotspot-регіоні встановлено наявність мікросателітних маркерів (МС) (англ. Simple Sequences Repeats, SSR) [17, 18].
В Ефіопському інституті сільськогосподарських досліджень (англ. Ethiopian Institute of Agricultural Research) способом зворотного схрещування за допомогою молекулярних маркерів розроблено й комерціалізовано толерантний до посухи високоврожайний сорт нуту ‘Geletu’ [19]. Так само виведено кенійські сорти нуту з ознакою «посухостійкість коренів» [20]. Через інтрогресію вищезгаданого QTL-hotspot-регіону методами маркер-опосередкованого добору (англ. marker-assisted selection, MAS) в Міжнародному науково-дослідному інституті сільськогосподарських культур для напівпосушливих тропіків (англ. International Crops Research Institute for the Semi-Arid Tropics) створено та згодом комерціалізовано толерантні до посухи ‘IPC L4-14’, ‘BGM 4005’ і ‘Pusa 102160’ [21].
У будь-якій селекційній MAS-програмі необхідною передумовою для скринінгу генотипів з цільовими (бажаними) ознаками є дослідження поліморфізму батьківських форм за генами, що відповідають за прояв цих ознак. Якщо маркери, які передбачено використовувати в MAS-програмі, не є поліморфними між батьківськими генотипами, подальший добір рослин, що несуть необхідні ознаки, неможливий у нащадках.
Мета досліджень – визначити поліморфізм мікросателітних локусів QTL-hotspot-регіону групи зчеплення 4, пов’язаного з толерантністю до посухи, у сортів нуту звичайного української селекції.
Матеріали та методика досліджень
Матеріалом для досліджень слугували сім сортів нуту української селекції (оригінатор – Селекційно-генетичний інститут – Національний центр насіннєзнавства та сортовивчення НААН України), а саме: ‘Буджак’, ‘Достаток’, ‘Маестро’, ‘Одисей’, ‘Пам’ять’, ‘Тріумф’ та ‘Ярина’.
Екстрагування та очищення ДНК виконували з розмелу 100 насінин ЦТАБ-методом [22]. Вимірювання концентрації та оцінювання якості екстрагованої ДНК здійснювали методом спектрофотометрії на спектрофлуориметрі «NanoDrop DeNovix DS-11 FX+» (DeNovix, США).
Полімеразну ланцюгову реакцію (ПЛР) проводили на термоциклері «QuantStudio 5 Real-Time PCR System» (Applied Biosystem, США) за такими температурно-часовими умовами: 1 цикл – 95 °C, 5 хв; 35 циклів – 95 °C, 30 с, 55 °C, 30 с, 72 °C, 1 хв; 1 цикл – 72 °C, 7 хв. Реакційна суміш містила 1× iTaq Universal Probes Supermix (BioRad, США), по 150 нМ прямого та зворотного праймерів (Metabion, Німеччина), 50 нг ДНК. Послідовності праймерів до МС локусів – згідно з [23].
Візуалізацію продуктів ампліфікації виконували методом горизонтального електрофорезу в 3%-му агарозному гелі (Agarose Wide Range, Serva, Німеччина) в 1× трис-боратному буфері (TBE buffer 10×, Serva, Німеччина) за напруженості електромагнітного поля 2 В/см протягом 4 годин. Для забарвлення гелів застосовували 0,5 мкг/мл розчину етидіум броміду (Sigma-Aldrich, США). Маркерами молекулярної маси слугували pUC19/Msp1 (HpaII) (ThermoScientific, США) та 50 bp DNA Ladder RTU (Cleaver Scientific, Велика Британія). Розміри фрагментів ампліфікації розраховували за допомогою програми «Image J» (ліцензія не потрібна) [24].
Кожен зразок аналізували у двох повтореннях ампліфікації. Контроль екстракції та безматричний контроль ампліфікації використовували для оцінювання контамінації.
Результати досліджень
Здатність екстрагованої ДНК до ампліфікації (тобто перевірку відсутності інгібіторів) оцінили методом ПЛР в режимі «реального часу», використовуючи TaqMan-систему праймерів та зонд до гена 18S rRNA еукаріот. Відмічено наявність ампліфікації (Ct 23–25) в усіх зразках нуту та її відсутність у контролі екстрагування й безматричному контролі. Отже, зразки ДНК нуту придатні для подальшого аналізу за МС локусами, коли псевдонегативні результати буде виключено та з’явиться можливість виявляти нуль-алелі локусів.
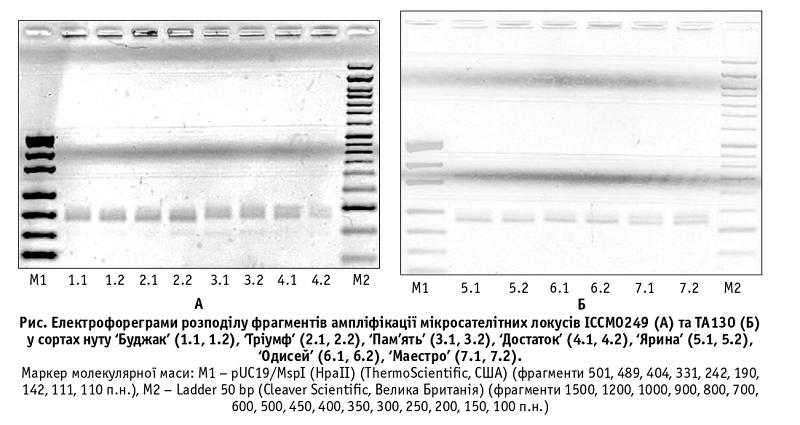
Для вивчення поліморфізму генотипів нуту використовували МС маркери QTL-hotspot-регіону, зокрема досліджено МС локуси ICCM0249, NCPGR127, TAA170, NCPGR21, TA130 та STMS11. На рисунку зображено електрофореграми розподілу фрагментів ампліфікації МС локусів.

У зразках контролю екстракції та безматричного контролю не відмічено ампліфікації за жодним із досліджених локусів.
МС локуси STMS11, NCPGR127, NCPGR21 виявилися неполіморфними в межах аналізованої вибірки сортів, зокрема детектовано по одному алелю кожного локусу розмірами 235, 200, 150 п.н. відповідно (табл.). Три MC локуси були поліморфними: для ICCM0249 і TAA170 ідентифіковано по два алелі, для TAA130 – два алелі та три алелі у сорту ‘Пам’ять’.
Порівнюючи дані проведеного дослідження щодо неполіморфних локусів з результатами, одержаними іншими дослідниками, можна зазначити, що локус STMS11 за аналізу 33 «desi» генотипів також був неполіморфним [23], розмір алеля відрізнявся і становив 220 п.н. Але цей локус виявився поліморфним: у вибірці з 36 генотипів встановлено три алелі розмірами 195–220 п.н. [25], зі 118 генотипів (115 місцевих сортів і селекційних ліній Ефіопії та три елітні сорти Індії) – 15 алелів розмірами 192–232 п.н. [26]. Локус NCPGR127 за аналізу 33 «desi» генотипів нуту був поліморфним [23] – ідентифіковано алелі 200 і 210 п.н., 118 генотипів [26] – 12 алелів розмірами 229–262 п.н. Вивчаючи локус NCPGR21, виявили шість алелів (135, 145, 150, 155, 160, 180 п.н.) серед 118 зразків (сортів) нуту з Європи (Україна, Молдова, Іспанія, Італія, Чехія), Азії (Індія, Узбекистан) та Америки (США, Канада) [27], 10 (розміри – 132–159 п.н.) – з-поміж 118 генотипів з Ефіопії та Індії [26], три алелі – за аналізу 33 «desi» генотипів (розміри – 150, 170, 190 п.н.) [23].
Щодо порівняння даних проведеного дослідження за поліморфними локусами, то під час аналізу 36 генотипів виявлено п’ять алелів локусу ICCM0249 розмірами 280–300 п.н. [25], а також алелі 180 і 200 п.н. за аналізу 33 «desi» генотипів [23]. У процесі вивчення 33 «desi» генотипів ідентифіковано чотири алелі локусу TAA170 (220, 240, 260 і 280 п.н.) [23], 36 генотипів – три алелі (200–220 п.н.) [25], 118 генотипів – 13 алелів (208–276 п.н.) [26]. Для локусу TA130 у дослідженні 33 генотипів встановлено три алелі (200, 220 і 230 п.н.) [23], 36 генотипів – чотири алелі (170–190 п.н.) [25], 118 генотипів – 22 алелі (224–289 п.н.) [26].
Очікувано кількість виявлених алелів підвищувалася зі збільшенням числа аналізованих генотипів: від трьох–п’яти, якщо досліджували 7, 33, 36 генотипів (це дослідження, 23, 25 відповідно), до 10–22 у разі вивчення 118 [27]. Діапазон розмірів виявлених у проведеному дослідженні алелів збігався з таким у [23, 26, 27] для локусів ICCM0249, TAA170, TA130, NCPGR127, NCPGR21. Алель 185 п.н. локусу STMS11, ідентифікований у сорту ‘Пам’ять’, також може бути присутній серед виявлених чотирьох алелів за аналізу 36 генотипів [25], але автори, вказуючи лише кількість і діапазон (чотири алелі розміром 170–190 п.н.), не уточнюють, які саме алелі детектували.
Отже, визначено п’ять типів алельних комбінацій проаналізованих МС л??????: ??? 1 ? окусів: тип 1 – у сортів ‘Буджак’ і ‘Тріумф’, 2 – ‘Достаток’ і ‘Маестро’, 3 – ‘Одисей’, 4 – ‘Пам’ять’, тип 5 – сорт ‘Ярина’. Алель розміром 185 п.н., відсутній в інших сортів української селекції з проаналізованої вибірки, виявлено в сорту ‘Пам’ять’.
Висновки
Установлено, що серед семи досліджуваних сортів нуту української селекції три мікросателітні локуси є неполіморфними, а саме: STMS11, NCPGR127 і NCPGR21. Ще три – поліморфними з двома алелями для локусів ICCM0249 і TAA170 і трьома для TAA130. Сорти розподілено за п’ятьма типами комбінацій алелів мікросателітних локусів ICCM0249, NCPGR127, TAA170, NCPGR21, TA130 і STMS11.
Майбутні дослідження будуть направлені на пошук кореляцій між алельним складом мікросателітних локусів, кількість яких збільшать, та ступенем толерантності до посухи розширеної вибірки генотипів нуту.
Результати цього дослідження можна рекомендувати використовувати в MAS-програмах підвищення толерантності до посухи, для характеристики корисних генетичних ресурсів і з метою розширення генетичної бази культивованого нуту.
Використана література
References
Таблиця
Алельний склад мікросателітних локусів генотипів нуту
|
Сорт |
Розміри алелів мікросателітних локусів, п.н. |
|||||
|
ICCM0249 |
TAA170 |
STMS11 |
NCPGR127 |
NCPGR21 |
TA130 |
|
|
‘Буджак’ |
180, 180 |
240, 260 |
235, 235 |
200, 200 |
150, 150 |
220, 230 |
|
‘Тріумф’ |
180, 180 |
240, 260 |
235, 235 |
200, 200 |
150, 150 |
220, 230 |
|
‘Пам'ять’ |
180, 200 |
260, 280 |
235, 235 |
200, 200 |
150, 150 |
185, 220, 230 |
|
‘Достаток’ |
180, 200 |
260, 280 |
235, 235 |
200, 200 |
150, 150 |
220, 230 |
|
‘Ярина’ |
180, 180 |
280, 280 |
235, 235 |
200, 200 |
150, 150 |
220, 220 |
|
‘Одисей’ |
180, 180 |
260, 260 |
235, 235 |
200, 200 |
150, 150 |
220, 220 |
|
‘Маестро’ |
180, 200 |
260, 280 |
235, 235 |
200, 200 |
150, 150 |
220, 230 |
UDC 577.21:575.22:581.6
Volkova, N. E.1,2*, Slishchuk, G. I.1,2, Zakharova, O. O.1, Marchenko, T. Yu.2, Sichkar, V. I.2, & Vozhehova, R. A.2
(2023). Analysis of chickpea (Cicer arietinum L.) genotypes by microsatellite loci of the QTLhotspotregion associated with drought tolerance. Plant Varieties Studying and Protection, 19(4), 00–00.
https://doi.org/10.21498/25181017.19.4.2023.291223
1Cotecna Ukraine Limited, 140A Lustdorfska doroha St., 65114, Odesa, Ukraine, *email: natalia.volkova@cotecna.com
2Institute of ClimateOriented Agriculture of the NAAS of Ukraine, 24 Maiatska doroha St., Hlibodarske village, Odesa region, 67667, Ukraine
Purpose. To determine the polymorphism of microsatellite loci of the QTLhotspotregion of linkage group 4, associated with drought tolerance in Ukrainian chickpea varieties. Methods. Extraction and purification of DNA from seedlings using the CTAB method; polymerase chain reaction; horizontal gel electrophoresis; determination of the size of amplification products using the “Image J” program. Results. Allelic combinations of microsatellite loci ICCM0249, NCPGR127, TAA170, NCPGR21, TA130, STMS11 of the QTLhotspotregion of linkage group 4 of the chickpea genome were established. It was found that the loci STMS11, NCPGR127, NCPGR21 were not polymorphic within the sample of varieties analyzed, one allele was detected for each locus; two alleles were detected for the loci ICCM0249 and TAA170 and three alleles for the locus TAA130, indicating their polymorphism. Conclusions. Microsatellite loci STMS11, NCPGR127, NCPGR21 are nonpolymorphic in seven Ukrainian chickpea varieties. Three loci are polymorphic with two alleles for ICCM0249 and TAA170 and three alleles for TAA130. According to the analysis of chickpea varieties, five types of allelic combinations of microsatellite loci ICCM0249, NCPGR127, TAA170, NCPGR21, TA130, STMS11 were established. An allele of 185 bp unique to the sample of cultivars studied was identified in the variety ‘Pamiat’.
Keywords: chickpea; drought tolerance; polymorphism; microsatellite locus; PCR.
Надійшла / Received 19.10.2023
Погоджено до друку / Accepted 24.11.2023